Entry information : EgrAPx-R (Eucgr.C01740 / Egrandis_v1_0.016593m)
| Entry ID | 8079 |
|---|---|
| Creation | 2011-02-04 (Christophe Dunand) |
| Last sequence changes | 2011-07-28 (Catherine Mathe) |
| Sequence status | complete |
| Reviewer | Qiang Li |
| Last annotation changes | 2012-06-19 (Qiang Li) |
Peroxidase information: EgrAPx-R (Eucgr.C01740 / Egrandis_v1_0.016593m)
| Name (synonym) | EgrAPx-R (Eucgr.C01740 / Egrandis_v1_0.016593m) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Class | Ascorbate peroxidase related [Orthogroup: APx-R001] | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Taxonomy | Eukaryota Viridiplantae Streptophyta Myrtaceae Eucalyptus | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Organism | Eucalyptus grandis [TaxId: 71139 ] | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Cellular localisation | N/D |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Tissue type | N/D |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Inducer | N/D |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Repressor | N/D |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Best BLASTp hits |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Gene structure Fichier |
Exons►
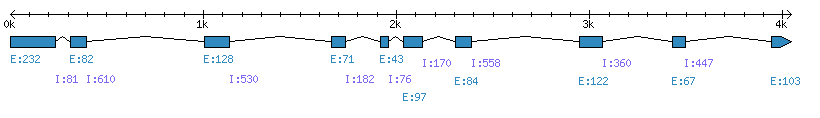
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Literature and cross-references EgrAPx-R (Eucgr.C01740 / Egrandis_v1_0.016593m)
| DNA ref. | Phytozome 12: scaffold_3 (29587959..29592001) |
|---|
Protein sequence: EgrAPx-R (Eucgr.C01740 / Egrandis_v1_0.016593m)
| Sequence Properties first value : protein second value (mature protein) |
|
||||||||||||
| Sequence Send to BLAST Send to Peroxiscan |
|
||||||||||||
| Remarks | Complete sequence from genomic. Incorrect prediction from Egrandis_v1_0.016593m where intron in PS is not predicted!!!. No EST. One other alternative transcript: Egrandis_v1_0.018459m (low identity for the PS when compared with PtAPx-R). | ||||||||||||
| DNA ► Send to BLAST |
|
||||||||||||
| CDS► Send to BLAST |
|
||||||||||||
